
精读分享│【Nature Genetics】:基于原始肺活量图的深度学习对慢性阻塞性肺疾病的推断识别出新的遗传位点并辅助风险模型的改良
英文题目:Inference of chronic obstructive pulmonary disease with deep learning on raw spirograms identifies new genetic loci and improves risk models
中文题目:基于原始肺活量图的深度学习对慢性阻塞性肺疾病的推断识别出新的遗传位点并辅助风险模型的改良
期刊:Nature Genetics(IF: 31.7)
单位:谷歌健康人工智能,美国东北大学电气与计算机工程系,布里格姆妇女医院的查宁网络医学部,印第安纳大学医学院医学与分子遗传学系,印第安纳大学医学院医学部心脏病学分部,布里格姆妇女医院肺病和重症监护医学分部,哈佛医学院
发表时间:2023年4月



摘要:
慢性阻塞性肺疾病(COPD)是全球第三大死因,具有高度遗传性。虽然目前临床上通过将肺功能综合指标的阈值来定义 COPD,但定量易感性评分能更有效地识别遗传信号。本研究利用深度卷积神经网络,基于有噪声的自我报告和国际疾病分类标签,从高维原始肺活量测定图中预测COPD病例-对照状态,并将模型预测结果作为易感性评分。这种基于机器学习(ML)的易感性评分不仅能准确区分 COPD 病例组和对照组,还能在无需领域专业知识的情况下预测COPD相关住院风险。此外,该评分与患者总体生存率和急性加重事件显著相关。基于ML易感性评分的全基因组关联研究不仅复现了已知的COPD和肺功能位点,还新发现了67个遗传位点。最后,该研究提供了一个通用框架:利用ML方法和基于医疗记录的标签,无需领域知识或专家整理即可显著提升疾病预测和基因组发现,以促进药物设计。
前言:
慢性阻塞性肺疾病(COPD)是一种以气流受限和持续气道炎症为特征的肺部疾病。据世界卫生组织最新评估,2019年COPD位列全球第三大死亡原因和第七大致残调整生命年(DALYs)诱因。尽管吸烟是主要危险因素,但COPD作为一种复杂且异质性的疾病,其发病机制涉及环境和遗传双重因素。在吸烟史相同的人群中,并非所有人都会患上COPD,这可能与遗传易感性有关。双胞胎研究和全基因组研究(GWAS)估计COPD的遗传率为40%~60%。根据全球慢性阻塞性肺疾病倡议(GOLD)标准,支气管扩张剂使用后的第一秒用力呼气量(FEV1)和用力肺活量(FVC)的比值<0.7即可诊断为COPD,而通过比较患者FEV1实测值与基于年龄、身高、性别和种族预测值(FEV1%预计值)可评估气流受限严重程度。
本研究假设使用原始的肺活量测定图构建COPD易感性评分(即 COPD 风险评分)可增强相关遗传位点的检出效能。相比常规汇总指标与固定阈值,肺活量测定图蕴含更多疾病风险与严重程度的评估信息。本研究扩展了基于机器学习(ML)的表型分析方法来预测COPD易感性——该方法通过ML模型构建合成表型进行遗传基础研究。既往研究表明,基于精确标签训练的ML表型模型能提供连续疾病风险度量(优于二元分类),从而提升关联分析效能。然而对某些难以收集的疾病而言,获取临床分级精确的疾病标签面临成本高、耗时长等挑战。相比之下,非完整医疗记录与自我报告的疾病标签虽准确性较低但更易获得。本研究证明了ML表型分析不一定需要精确的疾病标签,即使基于未经专家审核的噪声医疗记录疾病标签训练的ML模型,仍能提供具有生物学意义且具有临床预测价值的表型。
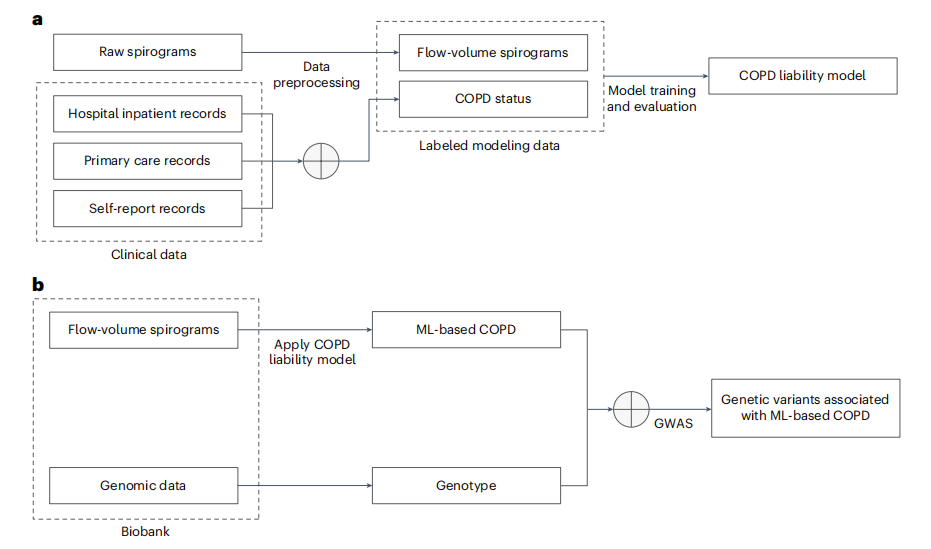
思维导图
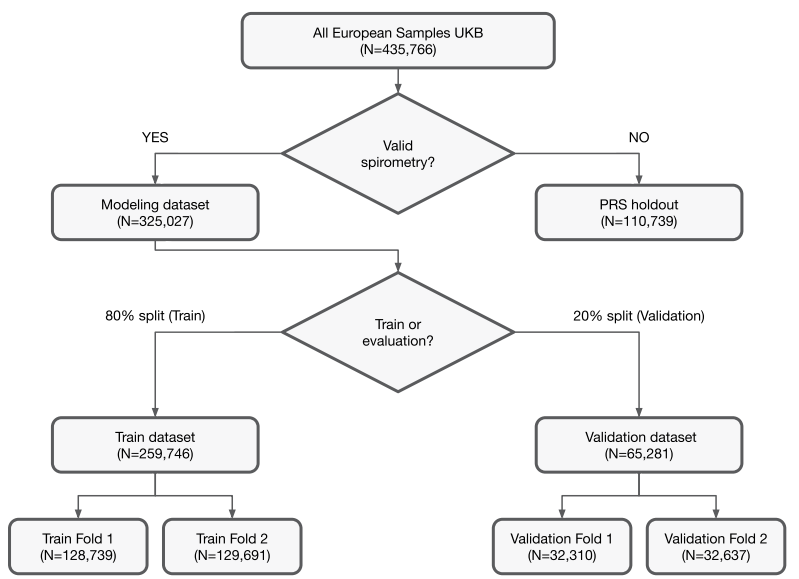
研究路线图
方法
数据来源
(1)UK Biobank 数据库
·325,027名欧洲血统受试者的肺功能图和基因型数据。
·随机分配以形成训练集和测试集,其中训练集包含80%的样本,用于模型的训练和优化;测试集包含20%的样本,用于模型的验证和性能评估。
(2)COPD 诊断
·COPD诊断:自我报告、住院病人ICD代码和初级保健或GP读取ICD代码
·Proxy-GOLD:基于FEV1/FVC < 0.7 和 FEV1预测值 < 80%
(3)肺功能图
·体积-时间曲线:描述时间依赖性的肺部呼吸的气体体积变化。
·流量-时间曲线:描述时间依赖性的呼吸气体流速的变化。
传统的肺功能诊断仅提取单一特征(如FEV1和FVC),而此研究利用了整个肺功能图的复杂信息。
2.标准定义
(1)GOLD标准:
·GOLD标准是用于诊断COPD的临床标准:FEV1/FVC< 0.7
·根据患者的 FEV1预测值,进一步分级严重程度:
轻度:FEV1预测值≥80%
中度:50%≤FEV1预测值< 80%
重度:30%≤FEV1预测值 < 50%
极重度:FEV1预测值 < 30%
(2)Proxy-GOLD标准:
·Proxy-GOLD是一种简化版的COPD诊断标准(使用单次吹气测试的FEV1和FVC数据),满足以下条件即为COPD病例:
FEV1/FVC < 0.7
FEV1预测值 < 80%
严重程度分级参考GOLD标准(尽管这些标签并非严格意义上的GOLD标签,因为仅使用了一次冲击且未进行支气管扩张,但它们仍能反映COPD状态)。
3.模型训练与验证
(1)模型架构:ResNet18-D
·使用改进版的一维卷积神经网络(CNN)架构,专门处理时间序列数据。
·输入:原始肺功能图(体积-时间和流量-时间曲线)。
·输出:COPD风险评分(连续值,反映个体患病风险的概率)。
(2)模型训练
·优化算法:使用Adam算法以端到端的方式优化网络,该算法通过自适应调整学习率来最小化模型预测概率与二元COPD状态标签之间的训练交叉熵损失,从而提高模型的收敛速度和稳定性。
·训练周期:模型最多训练1500个周期,以确保模型有足够的时间收敛到一个最优解。
·过拟合预防:采用提前停止耐心机制(50个周期),即如果在50个训练周期内模型在验证集上的性能没有提升,则停止训练,以防止模型过拟合训练数据。
(3)生成COPD风险评分
·风险评分是一个连续值,反映个体患COPD的风险大小。
·相比传统二分类(有病/无病),连续风险评分具有更高的分辨能力。
4.遗传关联研究
(1)目标:
利用COPD风险评分进行全基因组关联研究,发现与COPD相关的新遗传位点。
(2)表型定义:
研究分别使用以下表型变量进行GWAS:
·传统二分类COPD标签:1)Proxy-GOLD;2)COPD诊断
·深度学习生成的COPD风险评分:连续变量。
比较这两种表型变量的GWAS结果:
·能否发现更多的遗传位点。
·是否能更好地解释COPD的遗传基础。
(3)方法:
·将COPD风险评分作为表型变量。使用混合线性模型(MLM)进行GWAS,控制个体之间的遗传相关性和环境差异。
·调整混杂变量:年龄、性别、身高、年龄×性别(即年龄和性别的交互作用)、年龄×年龄和身高×身高、基因分型阵列和前15个遗传主成分(PCs)
·GWAS分析仅限于欧洲血统的个体。基因型过滤包括仅包含常染色体变体,其最小等位基因频率≥0.001,插值INFO得分≥0.8,以及哈迪-温伯格平衡(HWE)≥10⁻¹⁰。
·功能注释与验证:
功能富集:使用S-LDSC分析遗传位点在肺组织染色质开放区域的富集。
跨队列验证:
COPDGene队列:验证PRS在GOLD 2-4病例中的预测效能。
GBMI与SpiroMeta:验证新位点的效应方向一致性。
·模型评估指标:
AUROC/AUPRC:评估COPD诊断、住院和死亡的预测性能。
生存分析:Cox比例风险模型计算风险比(HR),Kaplan-Meier曲线展示生存差异。
SNP遗传力:S-LDSC估计h²=0.20(s.e.=0.01),表明责任评分具有显著遗传基础。
·敏感性分析:
条件分析:控制FEV1/FVC、FEV1等肺功能指标,排除其驱动效应。
DeepNull模型:校正年龄、性别与FEV1/FVC的非线性关系,验证结果稳健性。
结果
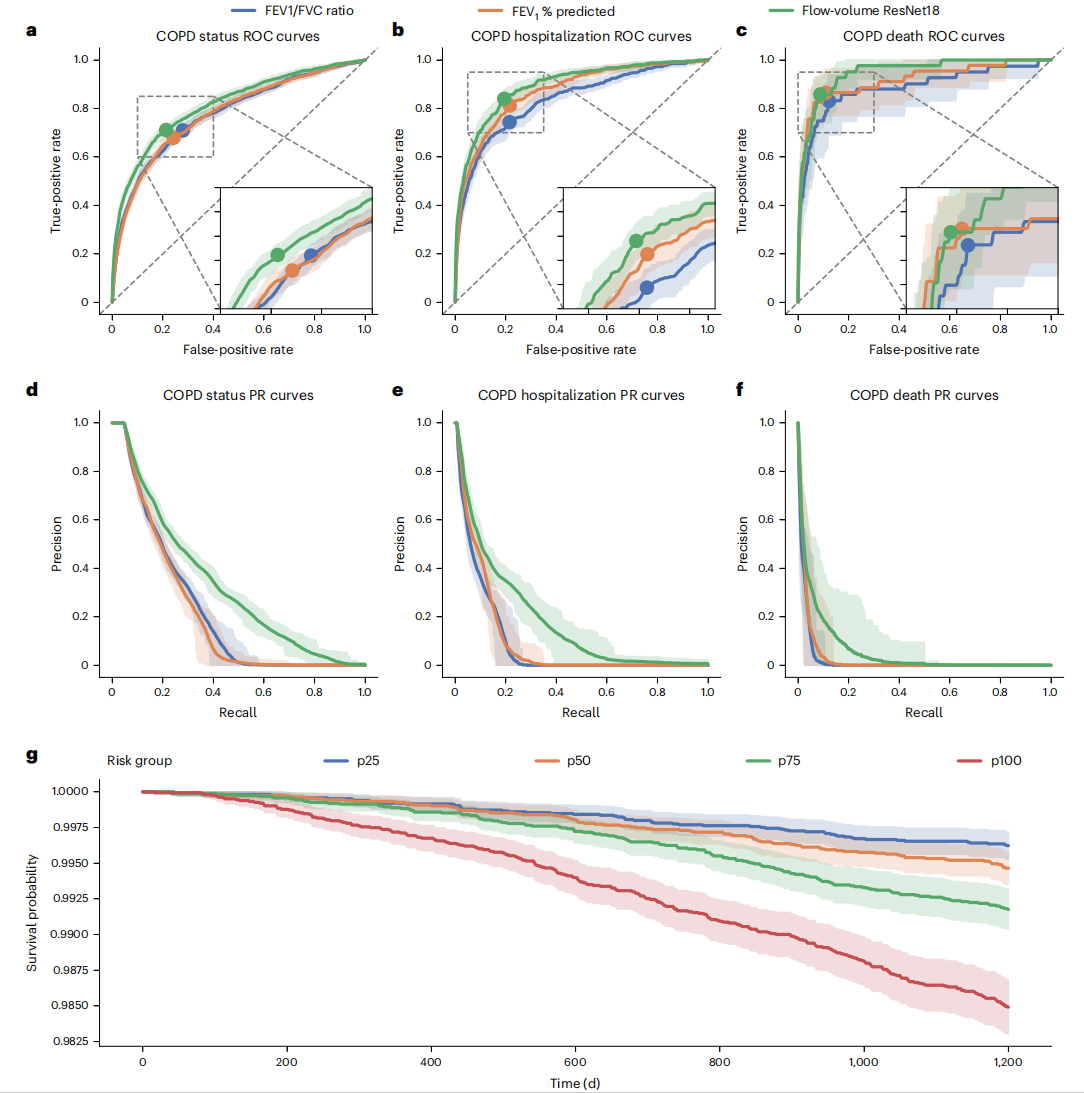
1、基于机器学习的COPD模型优于传统诊断方法
AUC对比结果(ROC曲线下面积越大越好):
(1)区分COPD病例与对照组:
ResNet18-D模型:AUC = 0.82
传统FEV1/FVC方法:AUC = 0.78
(2)预测未来COPD相关住院风险:
ResNet18-D模型:AUC = 0.89
传统FEV1/FVC方法:AUC = 0.87
(3)预测COPD相关死亡风险:
ResNet18-D模型:AUC = 0.95
传统 FEV1/FVC方法:AUC = 0.92
(4)总体生存分析:
使用Cox比例风险回归模型分析显示,每增加一个标准差的基于机器学习的COPD倾向评分,总体死亡的风险比(HR)为1.22(标准差=0.01;P≤2×10⁻¹⁶)。Kaplan–Meier曲线进一步表明,COPD风险较高的患者总体生存率下降更快。
2、新遗传位点的发现
(1)总体结果:
显著位点:356个独立位点(P≤5×10⁻⁸),其中67个为全新发现。
遗传力:SNP遗传力h²=0.20(s.e.=0.01),表明责任评分具有强遗传基础。
(2)新遗传位点的特征及生物学意义:
免疫功能相关基因:BCL11A、LTBR:与免疫功能和炎症反应相关。
肺组织发育与修复相关基因:BCL9、SFRP1:参与Wnt信号通路,调控肺组织发育和修复。
吸烟相关基因变异:CHRNA2、MAML3:与吸烟行为和尼古丁依赖性相关。
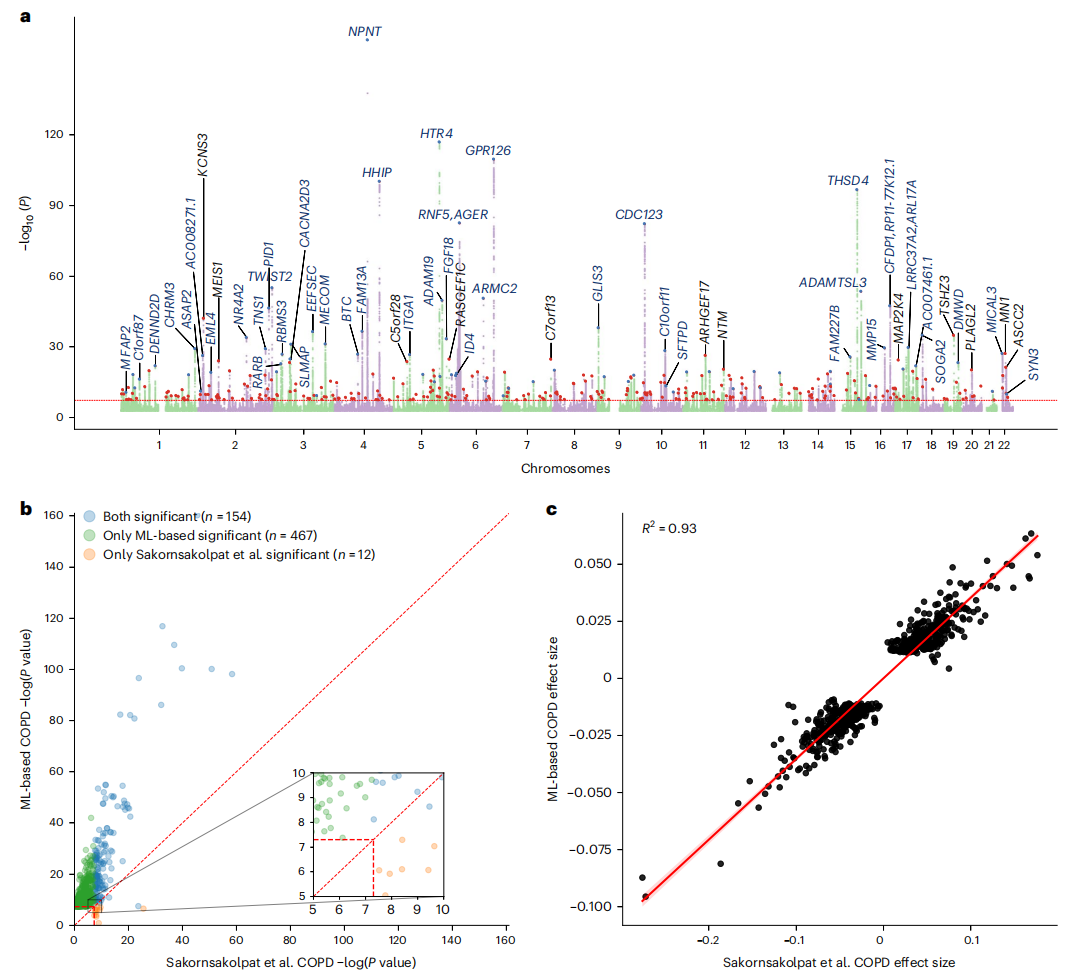
基于ML的COPD模型提高GWAS统计功效
(1)与Sakornsakolpat等(2019)的对比:
共享位点:154个位点在两项研究中均显著。
新发现:220个位点仅在本研究显著,12个仅在Sakornsakolpat中显著。
效应方向一致性:效应大小相关性R²=0.93(P<0.001),遗传相关性rg=0.90(s.e.=0.07)。
统计效力提升:相同位点的P值在本研究中普遍更低(例如NPNT位点P=1×10⁻²³ vs. 5×10⁻¹⁰)。
(2)条件分析验证:
控制FEV1/FVC、FEV1等指标后,仍检测到129个独立位点(P≤5×10⁻⁸),证明结果非肺功能驱动的假象。
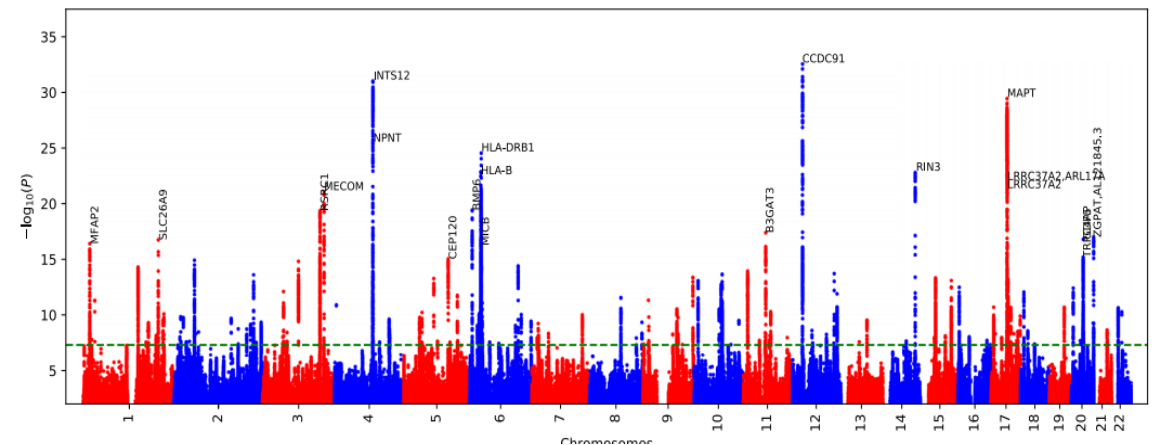
4、验证结果
基于机器学习的COPD评分的GWAS除已知的91个COPD基因位点外,还发现了265个新的COPD风险基因位点。
将67个新位点纳入验证数据集:GBMI(全球生物样本库荟萃分析倡议)、SpiroMeta和 ICGC(国际COPD遗传学联盟),这三个数据集均排除了来自英国生物样本库的样本。
在67个位点中,有38个位点发生了支持性的复制。此外,有6个位点发生了严格的复制。
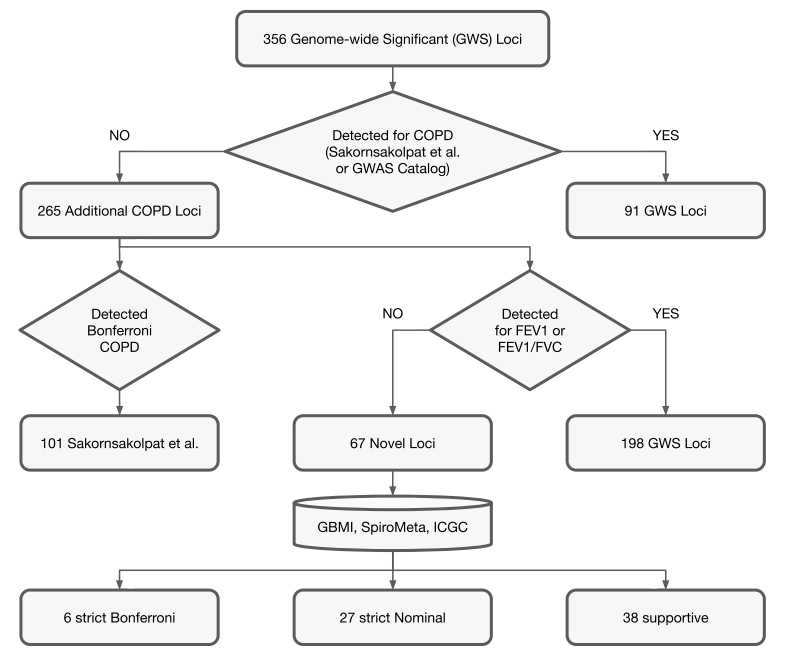
5、多基因风险评分(PRS)的评估
(1)PRS构建与比较:
研究者将GWAS中识别的遗传变异位点组合成简单的多基因风险评分(PRS),并在UKB和COPDGene数据集中评估其对高风险COPD病例的检测能力。结果显示,基于机器学习PRS在检测高风险COPD病例方面优于和Sakornsakolpat等人GWAS的PRS。
(2)不同数据集的表现:
在UKB中,基于机器学习的PRS在评估 COPD诊断、COPD相关住院和COPD相关死亡等任务上的AUROC分别为0.550、0.564和0.598,均高于基于COPD诊断 PRS和Sakornsakolpat等人的PRS。在COPDGene中,基于机器学习PRS与Sakornsakolpat等人PRS的性能相当,且均优于基于COPD诊断 PRS。
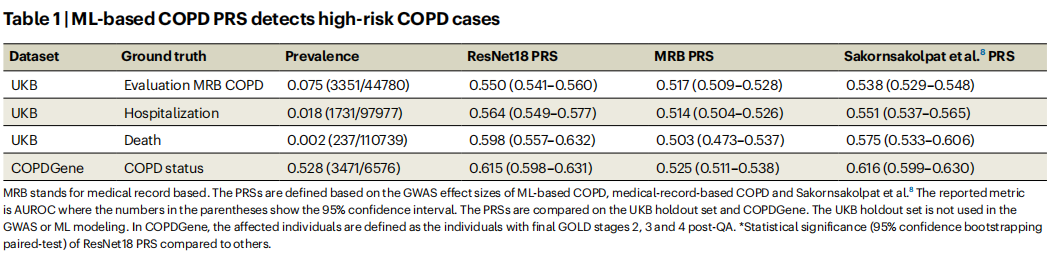
讨论
1.研究意义
(1)提高GWAS统计能力
连续风险评分提高了GWAS的统计能力,发现更多遗传信号,为COPD的遗传研究提供了新的突破,有助于深入理解COPD的遗传基础和疾病机制。
(2)揭示疾病异质性
新发现的位点可能揭示COPD的异质性病因,为个性化治疗和药物研发提供靶点,推动COPD的精准医学发展,提高治疗效果和患者预后。
(3)遗传学研究的拓展
为其他复杂疾病的遗传研究提供了新的思路和方法,具有广泛的应用前景。
2.机器学习模型的优势
(1)无需高质量标签
利用模糊标签(如自我报告)训练模型,依然能生成高效的COPD风险评分,降低了对高质量标签的依赖,提高了模型的实用性和可扩展性,尤其在数据标注困难的情况下具有重要意义。
(2)原始数据的充分利用
相比传统的FEV1/FVC指标,机器学习模型能够充分利用原始肺功能图的完整信息,捕捉更多与疾病相关的复杂模式,提高了模型的预测能力和对疾病异质性的识别能力。
(3)模型的泛化能力
模型在不同数据集和人群中表现出良好的泛化能力,具有较高的稳定性和可靠性,为模型的广泛应用和推广提供了基础。
3.研究局限
(1)种族局限性
仅分析了欧洲血统人群,可能限制结果的普适性,需要在其他种族和人群中进行验证,不同种族之间可能存在遗传和环境差异,影响模型的适用性。
(2)标签性能
训练标签噪声可能影响模型性能,虽然模型具有一定的抗噪声能力,但仍有改进空间,需要进一步优化标签处理方法,提高模型的准确性和可靠性。
(3)诊断标准偏差
未使用支气管扩张剂,可能与GOLD标准有差异,影响诊断的准确性,需要结合支气管扩张剂数据,进一步改进COPD诊断标准。

汇报人:庞文都
导师:赵宇 教授
审核:宋瑶、邱轲、任建君



