精读分享│【Nature Genetic】:海马和子域体积的跨族裔全基因组关联荟萃分析
英文题目:Cross-ancestry genome-wide association meta-analyses of hippocampal and subfield volumes
中文题目:跨族裔全基因组关联海马和子域体积的荟萃分析
期刊:Nature Genetic(IF: 30.8)
单位:天津医科大学总医院放射科、天津市功能影像重点实验室
发表时间:2023年12月



摘要:
海马体对于记忆、认知和神经精神疾病至关重要,其子区域在结构和功能上有所不同。海马体和子区域体积的全基因组关联研究主要在欧洲人群中进行;然而,其他族裔群体的代表性不足。在这里,我们对65,791名个体的海马体积和38,977名个体的子区域体积进行了跨族裔全基因组关联荟萃分析,其中包括 7,009 名东亚血统个体。我们针对44个海马性状识别出339个P < 1.13×10-9的变异-性状关联,其中包括23个新关联。尽管存在特定于祖先的关联,但常见的遗传变异对不同祖先的海马特征具有相似的影响。跨族裔荟萃分析提高了代表性不足人群中多基因评分的精细绘图精度和预测性能。这些基因变异被富集在 Wnt 信号传导和神经元分化,并影响认知、情绪和神经精神疾病。这些发现可能有助于深入了解海马体和亚区体积的遗传结构。
研究思路及方法:
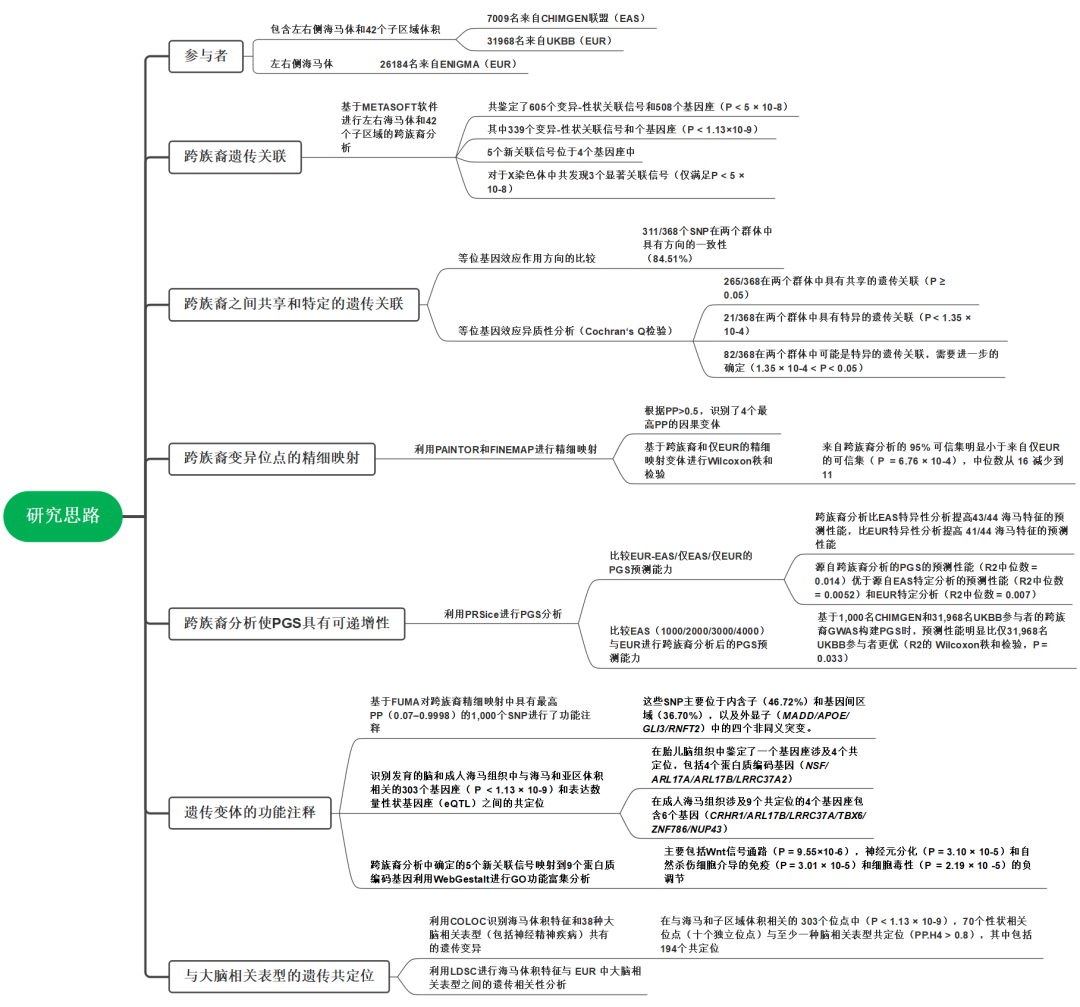
主要结果:
1、各队列中参与者
在本研究中,我们对左右大脑半球对称分布的44个海马和子区域结构的体积进行了跨族裔GWAS荟萃分析(图1a)。使用 GWAS 汇总统计数据对 EAS 和 EUR 参与者的这些特征进行了跨族裔分析。这些海马特征的 EAS-GWAS汇总统计数据(常染色体和 X 染色体)来自CHIMGEN 研究的7,009 名中国汉族参与者。对于左右海马体积,常染色体 EUR-GWAS 汇总统计数据是通过对来自英国生物银行 (UKBB)的 31,968 名 EUR 参与者的这两种表型的 GWAS和来自增强神经影像遗传学荟萃分析 (the enhancing neuroimaging genetics through meta-analysis consortia, ENIGMA) 联盟中26,814 名参与者的平均海马体积的 GWAS 进行荟萃分析获得的。对于海马子区域体积,常染色体 EUR-GWAS 汇总统计数据来自 31,968 名 UKBB 参与者。EUR-GWAS 伪常染色体区域 (PAR) 中 X 染色体变异的汇总统计数据来自 31,943 名 UKBB 参与者,非PAR 变异的汇总统计数据来自 31,954 名UKBB参与者。
2、跨族裔遗传关联
针对CHIMGEN、UKBB和ENIGMA中进行的GWAS共享的4,901,971个常染色体变异,我们使用固定效应模型对来自 CHIMGEN 的 7,009 名 EAS 参与者和来自UKBB和ENIGMA的58,782名欧洲参与者的左右海马体积进行跨族裔 GWAS荟萃分析。研究共鉴定了97个变异-性状关联(P< 5×10-8),其中52个关联满足P < 1.13 × 10-9。针对CHIMGEN-GWAS和UKBB-GWAS 共有的5,110,460个常染色体变异,我们对7,009名CHIMGEN和31,968名 UKBB 参与者的42个海马子区域体积进行了跨族裔荟萃分析,并确定了508 个变异-性状关联(P< 5×10-8),其中287个关联满足P< 1.13 × 10-9。其中在已识别的605个变异-性状关联(P < 5×10-8)中涉及518个性状相关基因座,其中137个关联在之前的研究中尚未报告。当合并44个海马性状的关联信号和基因座时,我们发现了126个独立的关联信号(102个基因座),其中27个关联信号和26个基因座从未被报道过与这些海马性状有关。339个变异-性状关联(P<1.13×10-9)包括23个新关联,涉及303个性状相关基因座(图1b)。当合并这 44 个海马性状的关联信号和基因座时,我们发现了56个独立的关联信号(44个基因座),其中包括5个关联信号和4个从未被报道过的任何这些海马性状的基因座(P < 5 × 10-8) 。第一个新关联信号(位于1号染色体处的先导SNP:rs823385;图1c)可能通过调节TRABD2B(包含2B的TraB 结构域)表达来影响Wnt蛋白结合和金属内肽酶活性。第二个新关联信号(位于9号染色体处的先导SNP:rs60371393;图1d )位于RABGAP1(RAB GTP酶激活蛋白1)的内含子中,涉及G蛋白偶联受体活性和小GTP 酶结合。第三个新信号(位于11号染色体处的先导SNP:rs12222581;图1e)包含MADD(MAP激酶激活死亡结构域)的两个非同义外显子变异(rs1051006和rs2290148),是一种神经保护基因。第四个新信号(位于15号染色体处的先导SNP:rs1390871;图1f)位于SEMA6D(信号蛋白6D)的内含子中,编码信号蛋白6D并调节轴突生长和引导。最后一个新信号(位于15号染色体处的先导SNP:rs9929688;图1g)被映射到编码锌指蛋白的ZNF423(锌指蛋白 423)。
针对CHIMGEN-GWAS和UKBB-GWAS共有的X染色体上的144,687个非PAR变异,我们对7,009名CHIMGEN参与者和31,954名UKBB参与者的 44个海马特征进行了跨族裔荟萃分析。针对CHIMGEN和UKBB共享的X染色体上的2,024个PAR变异,我们对7,009名CHIMGEN参与者和31,943名 UKBB参与者的这些性状进行了跨族裔GWAS荟萃分析。我们发现了三个显着的变异-性状关联(P < 5×10-8;补充表4),但是,当P<1.13×10-9时,没有一个存活下来。
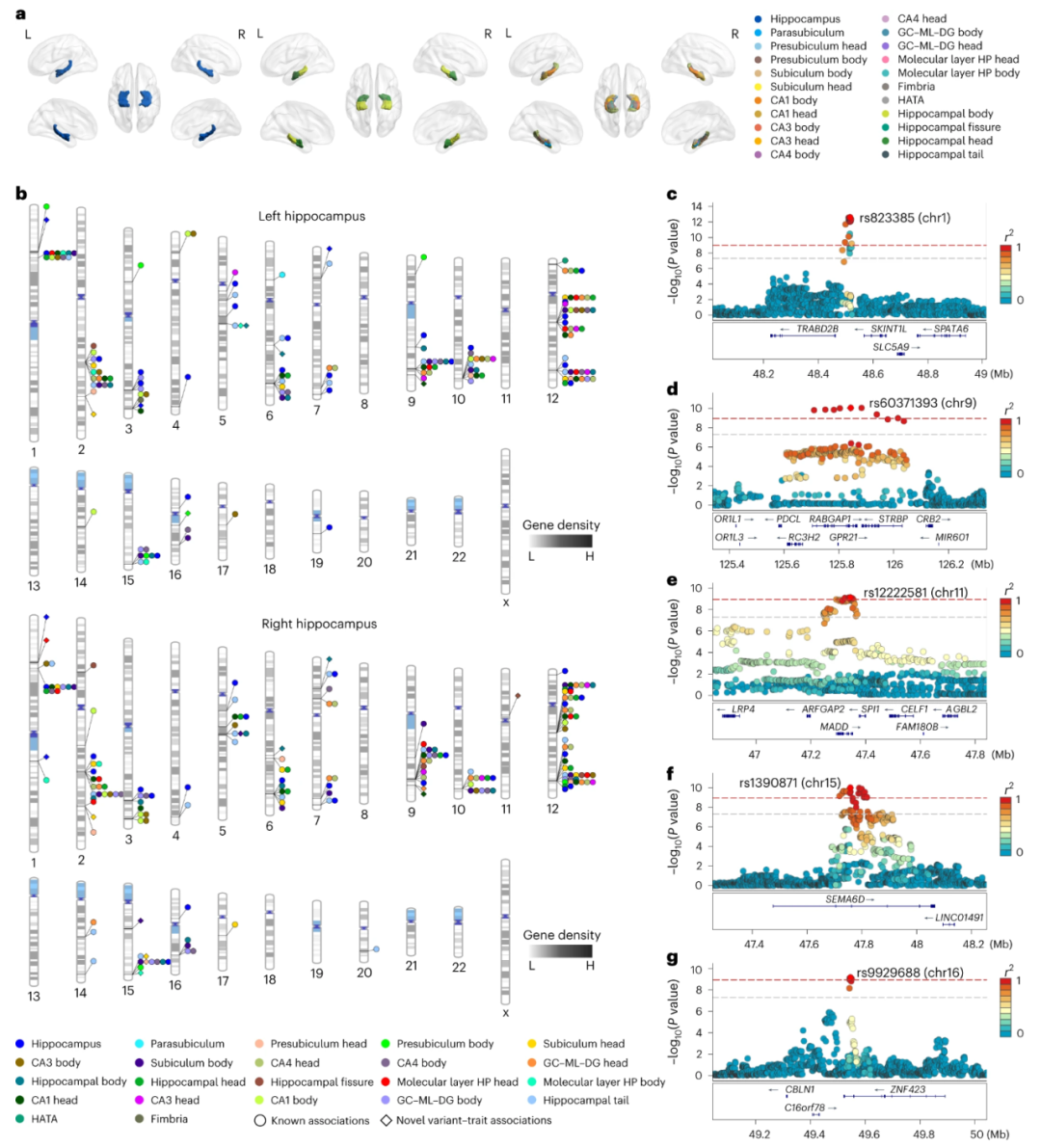
Figure 1. Cross-ancestry GWAS meta-analyses of hippocampal and subfield volumes
3、跨族裔之间共享和特定的遗传关联
由于尚未对非EUR人群的海马和子区域体积进行GWAS,因此EUR和EAS之间这些海马特征的跨族裔之间共享和特异性遗传关联仍然未知。在EUR和EAS之间的等位基因效应异质性分析中,我们纳入了所有在P< 1.13×10-9(44个海马性状)时存活的SNP包括海马体积的跨族裔GWAS荟萃分析(n = 65,791),EAS-GWAS(n= 7,009)或EUR-GWAS荟萃分析(n= 58,782),以及海马子区域体积中的跨族裔GWAS 荟萃分析(n= 38,977)、EAS-GWAS(n= 7,009)或 EUR-GWAS(n= 31,968)。排除重复关联和仅在一个群体中具有基因型数据的关联后,我们获得了368个变异-性状关联(P< 1.13×10-9)。我们比较了EAS和EUR之间每个先导SNP的作用方向,观察到两个群体之间的作用方向一致,有 84.51% 的关联性 (311/368)(图2a)。在等位基因效应异质性检验中,我们发现 EAS 和 EUR 之间存在 265/368 (72.01%) 跨族裔共享遗传关联,即没有任何等位基因效应异质性证据(Cochran's Q检验,P≥0.05)(图2b)。例如,先导SNP rs11245347(chr10)对EAS和EUR中的几个海马和子区域体积显示出显着影响,包括右侧海马尾部体积(图2c)。我们发现 21/368 (5.71%)跨族裔特异性遗传关联(图2b),即具有显着的等位基因效应异质性(Cochran's Q检验,P < 1.35×10-4,对368个变异性状关联进行Bonferroni校正)。例如,先导SNP rs7315280(chr12)在EAS和EUR之间的右侧海马尾部体积上显示出强烈的等位基因效应异质性(Cochran's Q检验,P = 2.14×10-8),且仅与EUR中的性状相关(图.2d)。应该指出的是,等位基因异质性可能是由群体之间真正不同的遗传效应驱动的,也可能是由仅影响标记变体的LD异质性驱动的。图2e显示了44个海马体和亚区体积中的跨族裔之间共享和特异性遗传关联的分布。在Cochran's Q检验中,我们还发现了82/368 (22.28%) 可能是显着的 (P< 0.05) 变异-性状关联,其类别需要在未来的研究中确定。
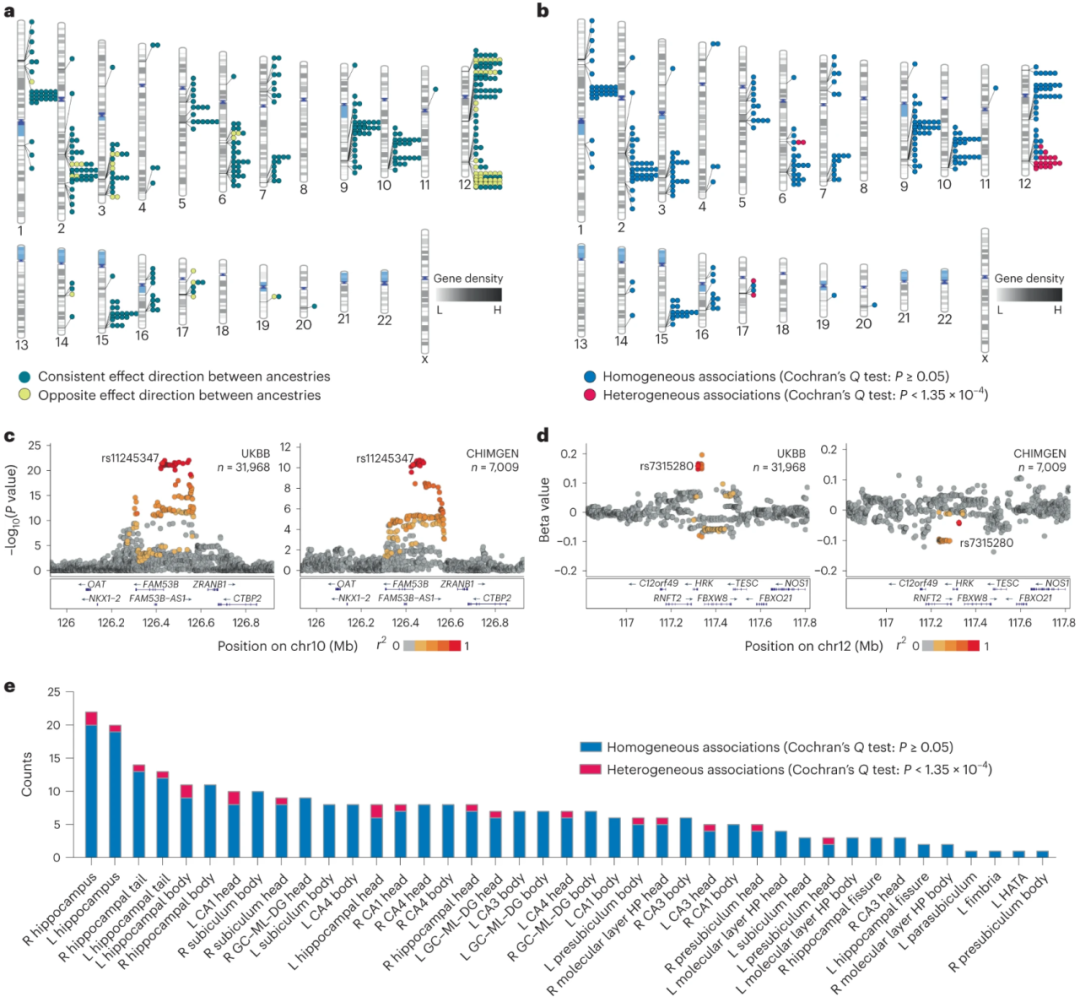
Figure 2. Ancestry-shared and ancestry-specific genetic associations of hippocampal and subfield volumes
4、跨族裔变异位点的精细映射
为了确定与海马和子区域体积相关的因果变异,以进行进一步的功能注释和调查,我们对7,009名EAS和58,782欧洲参与者的跨血统 GWAS 荟萃分析确定的303个性状相关位点进行了精细绘图,了解双侧海马体积和海马子区域(7,009名EAS 和31,968名EUR参与者)。使用基于马尔科夫链蒙特卡洛 (the Markov chain Monte Carlo, MCMC) 模型(允许多个因果变异)的概率注释积分器 (probabilistic annotation integrator, PAINTOR)进行精细映射,并通过基于鸟枪随机搜索的 FINEMAP确认所获得的结果,其允许的因果变异的最大数量为5。在这里,我们使用了样本量加权的LD参考集,该参考是根据建议的 CHIMGEN 和 UKBB 的插补后基因型数据构建的。此外,我们测试了LD参考集对跨祖先GWAS汇总统计的精细映射的影响,发现LD不匹配常常导致产生小的可信集,这可能被误认为是“有利的”的结果,是精细绘图研究中的一个潜在警告。PAINTOR 对303个性状相关基因座的精细作图结果如图3a所示。对于 303个性状相关位点,我们重新进行了EUR-GWAS汇总统计数据的精细映射,并对荟萃分析汇总统计数据进行了可疑位点分析(SLALOM)。PAINTOR 的后验概率(Posterior probability, PP)最高的 rs7315280 (chr12)(右侧海马体的PPPAINTOR = 0.9998和PPFINEMAP = 0.995;图3b)位于HRK(harakiri,BCL2 相互作用蛋白)的基因间区域,并调节HRK、FBXW8(包含8个F-box 和WD重复结构域)和TESC(tescalcin)。HRK对于神经元凋亡很重要,并且在基因型组织表达 (GTEx) 项目中在海马组织中表现出最高表达;FBXW8驱动海马神经元树突的生长和精细化并且TESC参与神经发育。rs7966895(齿状回 (GC–ML–DG) 头的右侧颗粒细胞和分子层;位于12号染色体,其中PPPAINTOR = 0.98 和 PPFINEMAP = 0.9992,图3c)被映射到HMGA2(高迁移率基团AT-hook 2),其在胎儿神经干细胞中高度表达并促进这些细胞的自我更新。rs6496265( 左海马体;位于15号染色体,PP PAINTOR = 0.94 和 PP FINEMAP = 0.97,图3d)被映射到ARRDC4(包含 4 的arrestin结构域),这是一种与能量产生相关的基因。
与之前的研究中使用一个推测的因果变体来比较精细映射精度的假设一致,在这里我们使用基于基因座内变体经PP排序后的联合概率生成的跨族裔和单族裔分析可信集的大小来进行比较,此外还使用这个假设来比较跨血统(7,009 EAS 和 58,782 EUR 参与者的双侧海马体积;7,009 EAS 和 31,968 EUR 参与者的海马子区域体积)和仅EUR-GWAS分析(58,782名参与者的海马体积;31,968名参与者的海马子区域体积)。在排除具有多个独立关联的基因座后,基于跨血统和仅欧洲人的GWAS 汇总统计数据中273/303个性状相关基因座,我们假设仅存在一种因果变异并使用 PAINTOR进行了精细作图。然后,我们使用 Wilcoxon 秩和检验(P <0.05)来研究与仅EUR相比,跨族裔荟萃分析的精细映射是否可以减少这些性状相关基因座95%的可信集数量。我们发现,来自跨族裔分析的 95% 可信集明显小于来自仅EUR的可信集(P = 6.76×10-4),中位数从 16 减少到 11(图3e)。198/273 (72.53%) 个性状相关位点的精细映射分辨率得到了提高,最精确的精细映射数量(95% 可信集中的一个变体)从仅EUR中的6个增加到了在跨族裔荟萃分析中的12个。一个例子是在左海马体中以 rs6496265 (chr15) 作为先导SNP的基因座(图3f),其中 95% 可信集中的因果 SNP 数量从仅EUR中的 9 个变为跨族裔精细映射中的 1 个。
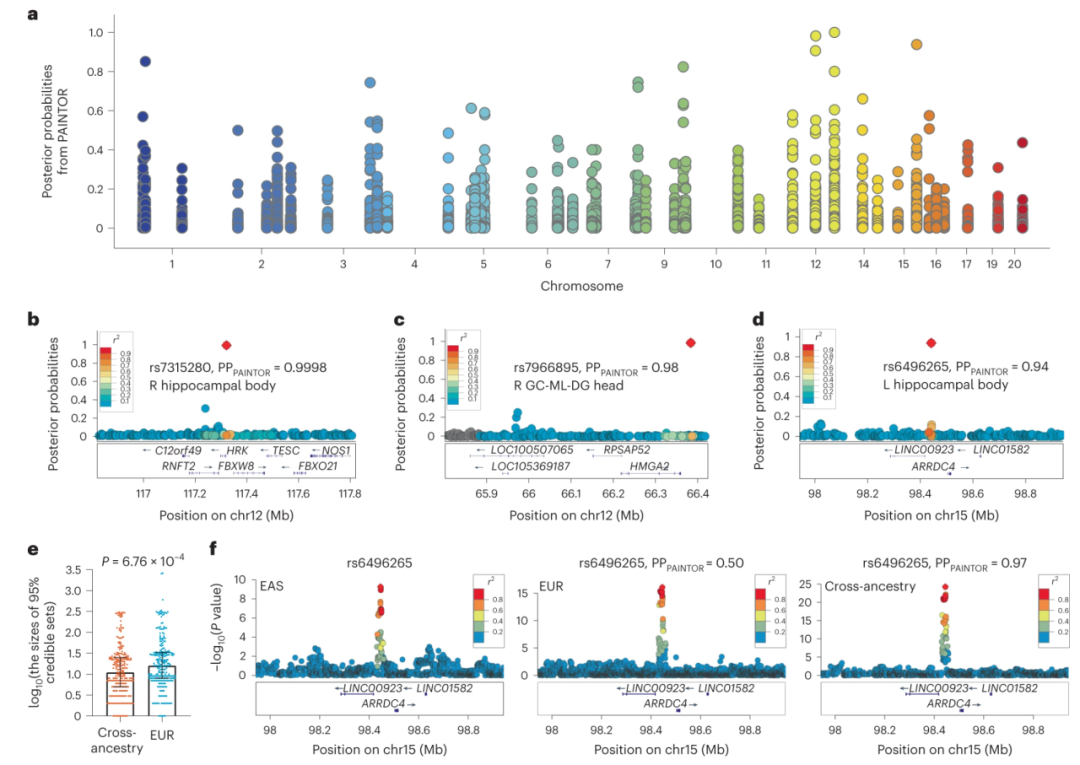
Figure 3. Cross-ancestry analysis improves fine-mapping resolution.
5、跨族裔分析使PGS具有可递增性
到目前为止,所有针对海马和子区域体积的大规模GWAS都是在欧洲个体中进行的;然而,仅基于欧洲个体构建的PGS在预测非欧洲人群个体的特征时往往表现不佳。在这项研究中,我们系统地评估了基于不同族裔的个体的不同组合构建的PGS的适用性,以预测代表性不足人群(EAS)个体的海马特征。在这些分析中,CHIMGEN参与者根据磁共振(MR)扫描仪的类型分为以下两组:5,010名参与者,其 MRI 数据由相同类型的扫描仪(GE MR750)采集,参数被视为基础数据集在PG分析中,其余1,999名参与者的MRI数据由10种类型的MR扫描仪获取,被视为目标数据集(补充图5)。我们比较了基于以下三个GWAS汇总统计数据构建的 PGS 的预测性能:(1)36,978名参与者(5,010名来自 CHIMGEN,31,968名来自UKBB)的跨族裔GWAS荟萃分析,(2)31,968名 UKBB中的仅EUR-GWAS和 (3) 5,010名CHIMGEN参与者中的仅EAS-GWAS。由于我们对PGS对代表性不足人群中海马特征的预测性能感兴趣,因此我们只纳入了来自CHIMGEN的1,999名EAS参与者作为所有PGS分析的目标数据集。跨族裔分析比EAS特异性分析提高43/44 海马特征的预测性能,比EUR特异性分析提高 41/44 海马特征的预测性能(图4a)。我们发现,源自跨族裔分析的PGS的预测性能(R2中位数 = 0.014)优于源自EAS特定分析的预测性能(R2中位数 = 0.0052;Wilcoxon秩和检验:P = 9.66×10−13)和EUR特定分析(R2中位数 = 0.007;Wilcoxon秩和检验:P = 1.26×10-8;图4b)。
我们进一步评估了来自跨族裔荟萃分析PGS的预测性能,其中包括基础数据集中不同数量(1,000、2,000、3,000和4,000)的 EAS 参与者,这些参与者是根据5,010个CHIMGEN中的随机抽样提取的参与者,发现EAS参与者的加入可以提高PGS的预测性能(图4c,d)。当我们基于1,000名CHIMGEN和31,968名UKBB参与者的跨族裔GWAS构建PGS时,预测性能明显比仅31,968名UKBB参与者更优(R2的 Wilcoxon秩和检验,P = 0.033;图4d)。随着跨族裔荟萃分析中 CHIMGEN 参与者数量的增加,构建的 PGS 的预测性能逐渐提高。总而言之,这些结果表明基于跨族裔GWAS 结果构建的 PGS 可以通过适度的增加来自该人群个体样本来提高对代表性不足人群中个体特征的预测性能。
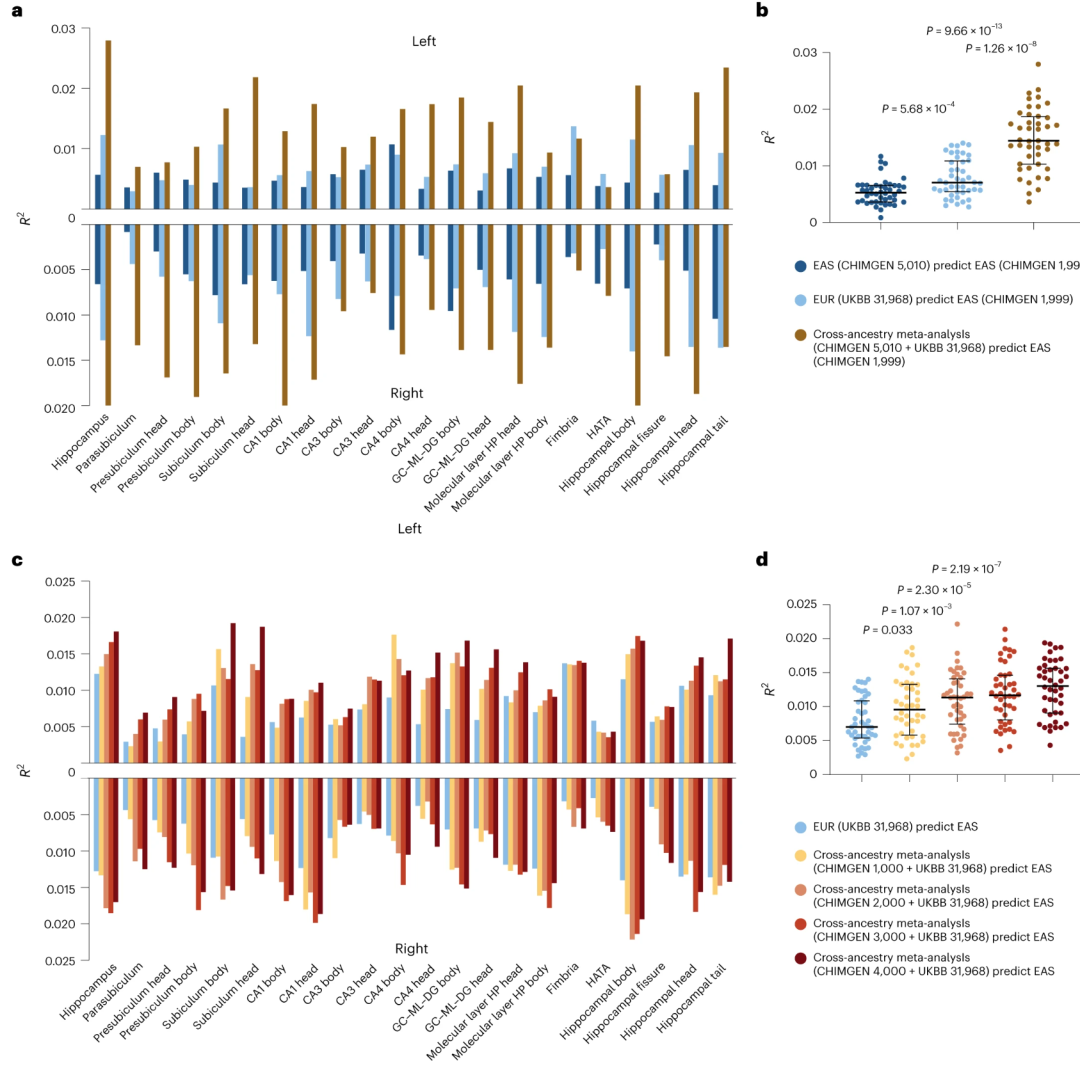
Figure 4. Prediction performance for hippocampal volumetric traits using PGSs constructed by different schemes.
6、遗传变体的功能注释
为了探索与海马和子区域体积相关的遗传变异的功能信息,我们使用基于网络的综合平台(功能映射和注释,FUMA)对跨族裔精细映射中具有最高 PP(0.07–0.9998)的1,000个SNP进行了功能注释。使用注释变异 (ANNOVAR)和组合注释依赖性缺失(combined annotation-dependent depletion, CADD) 评分研究了这些 SNP 的功能信息。在这1000个SNP中,963个被包含在FUMA中,959个被包含在ANNOVAR中,这959个SNP主要位于内含子(46.72%)和基因间区域(36.70%)(图5a)。我们还确定了外显子中的四个非同义突变,包括通过跨族裔荟萃分析确定的一个突变(MADD中chr11处的rs2290148)(P< 1.13×10-9)。AD 病理过程的海马体中MADD的表达减少,从而导致神经元细胞死亡。chr19处的rs429358(CADD score = 12.64)是APOE(载脂蛋白 E)的错义突变,与海马体积和AD相关。另外两个错义突变是GLI3(GLI家族锌指3)中的rs929387(chr7)和RNFT2(环指蛋白,跨膜2)中的rs903775(chr12),其中GLI3与大脑发育相关。具有高CADD(18.27)和高PP(0.64)的rs7030607(chr9)位于ASTN2(astrotactin 2)的内含子中,ASTN2(astrotactin 2)编码神经元蛋白astrotactin 2,与神经元迁移和神经发育障碍相关。
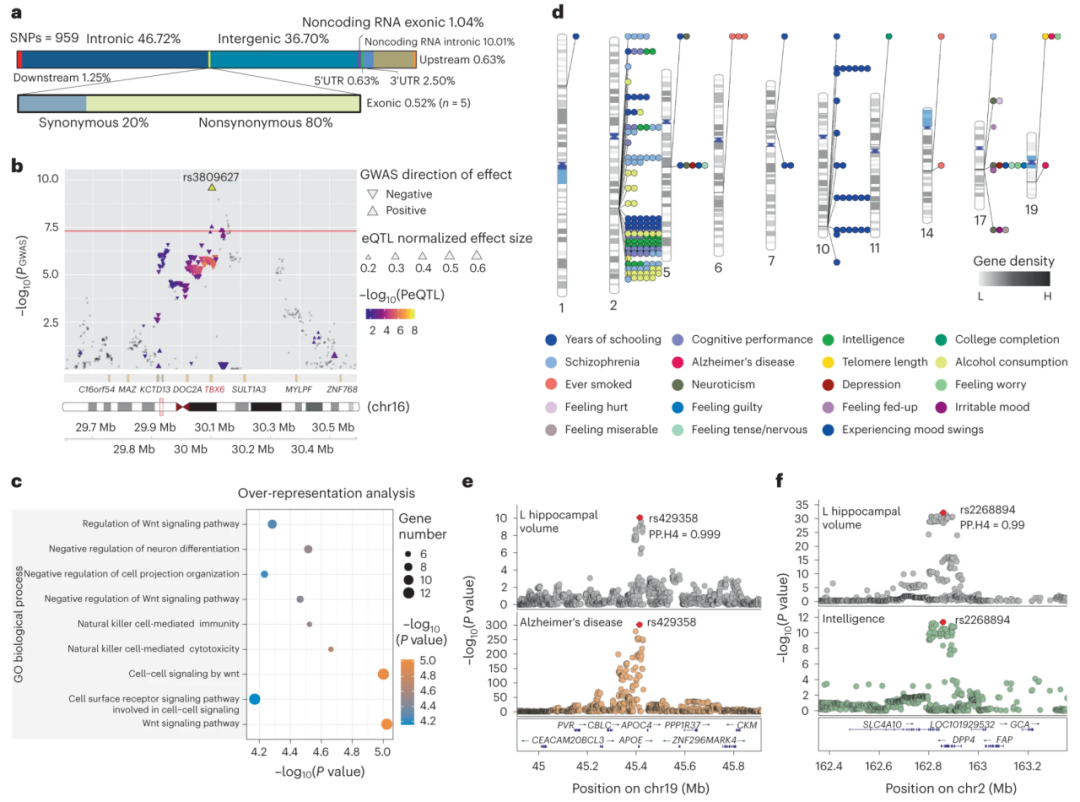
Figure 5. Functional annotations of genetic variants associated with hippocampal and subfield volumes
Coloc被用于识别发育的脑和成人海马组织中与海马和亚区体积相关的303个基因座( P < 1.13 × 10-9)和表达数量性状基因座(eQTL)之间的共定位。当 PP.H4(共享因果变异的 PP)阈值 > 0.8时,我们对于胎儿脑组织涉及一个基因座鉴定了4个共定位,涉及4个蛋白质编码基因(NSF,N-乙基马来酰亚胺敏感因子,囊泡融合(vesicle fusing)ATP酶;ARL17A,ADP核糖基化因子类GTP酶17A;ARL17B,ADP 核糖基化因子类GTP酶17B;以及LRRC37A2,富含亮氨酸的重复序列包含37成员A2),以及在成人海马组织涉及9个共定位的4个基因座包含6个基因(CRHR1,促肾上腺皮质激素释放激素受体1;ARL17B,LRRC37A,亮氨酸的富含重复序列包含37A;TBX6,T-box转录因子6;ZNF786,锌指蛋白786;和NUP43,核孔蛋白43)。例如,左侧 (PP.H4 = 0.999) 和右侧 (PP.H4 = 0.996) 海马体积均显示出与海马TBX6表达的共定位,这在抑制神经发育中具有重要作用。在左侧海马体积的共定位中(图5b),chr16处的rs3809627具有最大的PP,其中有效等位基因(A)显示海马TBX6表达减少和海马体积增加。
根据位置,FUMA中包含的963个SNP被映射到113个蛋白质编码基因,其中跨族裔分析中确定的5个新关联信号并被映射到9个蛋白质编码基因。我们使用 WebGestalt 进行统计过度表征分析,以确定这些基因被富集的生物过程基因本体 (GO) 术语。这些基因被富集到9个GO生物过程术语(qc<0.05,Benjamini-Hochberg错误发现率(BH-FDR)校正;图5c),主要包括Wnt信号通路(P= 9.55×10-6),神经元分化(P= 3.10×10-5)和自然杀伤细胞介导的免疫(P = 3.01×10-5)和细胞毒性(P = 2.19×10-5)的负调节。在跨族裔分析中确定的新关联信号映射的九个基因中,TRABD2B和SEMA6D分别富集在Wnt信号通路和神经元分化。
7、与大脑相关表型的遗传共定位
Coloc被用于识别海马体积特征和38种大脑相关表型(包括神经精神疾病在内的共38种表型和77个GWAS)共有的遗传变异。在与海马和子区域体积相关的 303个位点中(P< 1.13 ×10-9),70个性状相关位点(十个独立位点)与至少一种脑相关表型共定位(PP.H4 > 0.8),其中包括194个共定位(图5d)。在神经精神疾病的共定位分析中,左侧海马体积显示与 AD 共定位于 rs429358(chr19;图5e),APOE的外显子变体。精神分裂症显示出与 29个海马特征的共定位,主要位于具有先导SNP rs6432708(chr2)的基因座,该基因座也与智力 (图5f)、受教育年限、认知表现和饮酒量共定位。位点中的共享 SNP 被映射到SLC4A10(溶质载体家族4成员10)和DPP4(二肽基肽酶4)。SLC4A10编码 Na+偶联的HCO3-交换器,该交换器在CA3锥体细胞中含量丰富,可调节神经元兴奋性和突触短期可塑性。DPP4在海马体中高表达并与认知障碍相关。认知相关表型显示出与6个位点的35个海马特征的共定位。例如,多年的学校教育表明,在具有先导SNP rs4962691(chr10)的基因座上,26个海马特征存在共定位,该基因座被映射到涉及Wnt信号通路的FAM53B(具有序列相似性的家族 53 成员 B)。我们还发现海马体积特征与情绪和神经质的遗传共定位。补充表14中提供了这些海马体积特征与 EUR 中大脑相关表型之间的遗传相关性。
汇报人:舒涛 编辑:陈秋蓉 审核:任建君、吴桂儀



