精读分享│【Nature Genetics】:The evolutionary dynamics of extrachromosomal DNA in human cancers
英文题目:The evolutionary dynamics of extrachromosomal DNA in human cancers
中文题目:人类癌症中染色体外DNA(ecDNA)的动态进化
期刊:Nature Genetics(IF=31.7)
单位:美国斯坦福大学医学院
时间:2022年9月


摘要
Oncogene amplification on extrachromosomal DNA (ecDNA) is a common event, driving aggressive tumor growth, drug resistance and shorter survival. Currently, the impact of nonchromosomal oncogene inheritance—random identity by descent—is poorly understood. Also unclear is the impact of ecDNA on somatic variation and selection. Here integrating theoretical models of random segregation, unbiased image analysis, CRISPR-based ecDNA tagging with live-cell imaging and CRISPR-C, we demonstrate that random ecDNA inheritance results in extensive intratumoral ecDNA copy number heterogeneity and rapid adaptation to metabolic stress and targeted treatment. Observed ecDNAs benefit host cell survival or growth and can change within a single cell cycle. ecDNA inheritance can predict, a priori, some of the aggressive features of ecDNA-containing cancers. These properties are facilitated by the ability of ecDNA to rapidly adapt genomes in a way that is not possible through chromosomal oncogene amplification. These results show how the nonchromosomal random inheritance pattern of ecDNA contributes to poor outcomes for patients with cancer.
染色体外DNA(ecDNA)上的癌基因扩增是一种常见现象,癌基因的扩增驱动了肿瘤的快速生长、耐药性的增加以及患者生存期的缩短。目前,我们对非染色体上癌基因的遗传,即随机同源性遗传对肿瘤细胞的影响,仍然知之甚少。另外,ecDNA对体细胞变异和选择的影响也不明确。本文通过整合随机分裂的理论模型、无偏图像分析、基于CRISPR的ecDNA标记与活细胞成像以及CRISPR-C技术,证明了随机的ecDNA遗传会导致肿瘤内部ecDNA拷贝数的广泛异质性,并促使肿瘤细胞迅速适应代谢应激和靶向治疗。进一步观察表明,ecDNA有利于肿瘤细胞的存活和生长,其数量可以在一个细胞周期内发生变化。ecDNA可以预测肿瘤的侵袭性,因为ecDNA能够快速调整基因组,而这种快速适应的能力是染色体上的癌基因没有的。结果表明,ecDNA的随机遗传模式对癌症患者的不良预后有重要贡献。
研究背景:
(1)ecDNA的背景知识:
1. 定义:
ecDNA(extrachromosomal DNA):是指游离在细胞核内染色体以外的无着丝粒、无端粒的环状DNA,大小通常在1-3Mb,具有完整的基因以及调控区域,光学显微镜下能看到。
2. ecDNA与肿瘤的关系:
从2013年开始,越来越多证据证明,ecDNA在肿瘤细胞中广泛存在。
从2017年开始,有研究发现,癌基因的扩增在ecDNA上广泛存在,在天然染色体上的扩增较少。由于ecDNA没有着丝粒,不会随着细胞分裂均分,所以癌基因的拷贝数能随着细胞分裂很快在一些癌细胞中增加,实现癌基因在细胞中的快速扩增,比常染色体上的癌基因扩增速度快很多。并且有研究认为,癌基因拷贝数增加是导致癌基因表达上调的重要因素。
这说明ecDNA上癌基因的扩增是造成肿瘤内细胞异质性的关键,也是肿瘤进化和耐药性产生的关键。
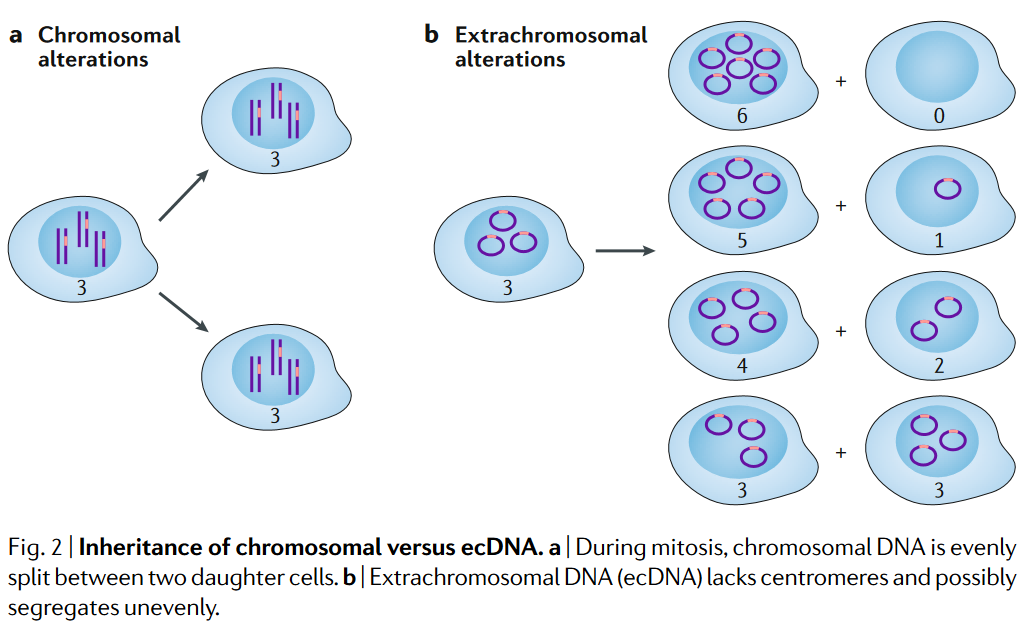
这篇研究的主要内容,就是通过计算机算法和实验,在各种肿瘤细胞系和患者样本组织中验证上述观点:ecDNA能促进肿瘤异质性、肿瘤进化和产生耐药性。
(2)该研究的背景:
1. 达尔文进化理论(肿瘤细胞遗传变异、自然选择),一直是解释肿瘤发生发展和产生适应性的理论基础。传统的肿瘤克隆进化理论认为癌基因是随着细胞分裂过程中,染色体的复制,着丝粒的分裂均匀分配到两个子细胞中的。
2. 然而,有一些现象难以只用传统的遗传进化模型来解释,比如:
①肿瘤内基因拷贝数的高度异质性(理论上按照自然选择,应该保留优势细胞群体)。
②肿瘤的快速适应性(染色体的遗传无法做到基因拷贝数的快速变异)。
③肿瘤的耐药突变速度太快(耐药突变的产生速度比模型预测的快)。
3. ecDNA的快速扩增能解释这些现象,因为它没有着丝粒,能解释肿瘤内细胞高度异质性、癌基因拷贝数快速变异,耐药性的快速产生的原因。但这些假设还没有得到证实。
因此,该研究整合了多种技术,包括计算机算法和数学模型、FISH+图像分析、CRISPR/Cas9基因编辑、活细胞成像、以及肿瘤患者的纵向分析,深入理解ecDNA的遗传机制及其功能性影响。
研究方法和思路:

(1)荧光原位杂交(FISH)+显微成像分析:
固定有丝分裂中期的细胞——用FISH探针标记靶基因——FISH荧光位点的量化,统计ecDNA的拷贝数(ImageJ是一个图像处理软件)。
(2)计算机模拟(通过Gillespie算法等,模拟肿瘤细胞群生长过程中单个细胞中ecDNA的数量变化)
1.起始状态设置
①模拟细胞系的动态增殖:从含有n个ecDNA拷贝的单个细胞开始(n是这个细胞系的平均细胞ecDNA拷贝数),因为细胞系在实验室中长期培养之后ecDNA的拷贝数会比较高。
②模拟患者样本的动态增殖:从含有1个ecDNA拷贝的单个细胞开始,因为患者样本中的ecDNA拷贝数通常比较少。
2.环境条件设置:可以根据环境压力来设置参数s,s=1代表中性选择环境,s>1代表正向选择环境,0<s<1代表负向选择环境。
3.终点设置
①细胞系:细胞数量达到106个时。
②患者样本:细胞数量达到1011个时。
输出结果:得到该细胞群中每个细胞的的ecDNA拷贝数,ecDNA拷贝数在细胞群中的分布。
(3)数学模型:
该研究构建了一个数学模型,来模拟各种条件下肿瘤细胞群中ecDNA的扩增,这个模型的构建基于5个假设:
①ecDNA随机进入两个子细胞
②肿瘤细胞群的细胞数量呈指数增长
③ecDNA的复制频率和细胞分裂一致
④肿瘤细胞群是由1个携带1个ecDNA的细胞开始的
⑤ecDNA的拷贝数丢失是不可逆的
数学模型:
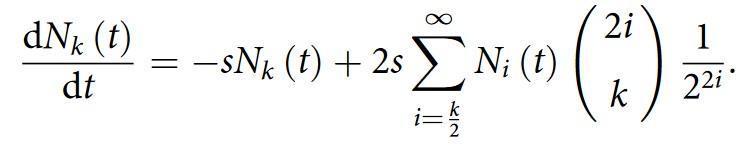
这个方程中k和t是自变量,t是时间(连续变量),k是单个细胞中携带的ecDNA的数量(离散变量),Nk(t)是因变量,代表t时刻有k个ecDNA的细胞数量,s是一个可调节的系数,可以根据环境压力进行调整(s=1代表中性选择环境,s>1代表正向选择环境)
(4)CRISPR-Cas9
构建含有目标基因的ecDNA。
(5)微滴式数字PCR(ddPCR):用于准确检测样本中的ecDNA的数量
ddPCR,是继一代普通PCR、二代荧光定量PCR之后的第三代PCR技术,其原理是将一个样本分充分稀释,分配到不同的反应单元,每个单元包含少于或等于一个拷贝的目标分子(某特定片段,在本文中指ecDNA),在每个反应单元中进行单独、平行的PCR反应,扩增结束后对各个反应单元的荧光信号进行统计(有荧光就记为1),以实现绝对定量及稀有等位基因的检测。
研究结果:
(1)实验验证ecDNA随机分配进入子细胞的这一观点
问题:ecDNA是否随母细胞有丝分裂随机进入两个子细胞(或者我们是否可以观察到癌基因特异性差异)?
假设:ecDNA是随机进入两个子细胞的,各种子细胞的ecDNA数量是呈正态分布的(fig.1a)
方法1:FISH结合图像分析技术,观察并统计子细胞中ecDNA的数量:使用FISH探针检测位于这些ecDNA上的扩增癌基因,并通过Aurora B激酶免疫染色来识别晚期有丝分裂中的子细胞,验证假设。
样本:
选择了多种人肿瘤细胞系(前列腺癌PC3、结肠癌COLO320DM、胃癌SNU16、胶质母细胞瘤GBM39-EC、神经母细胞瘤CHP212等),每个细胞系选100对子细胞,观察子细胞ecDNA的数量和分布(Extended Data Fig.2、fig.1b,c)。
结果:
1.不同肿瘤细胞内ecDNA上的癌基因不同,有些肿瘤细胞不止一种ecDNA(Extended Data Fig.2a)
2.FISH图像观察细胞有丝分裂晚期ecDNA进入2个子细胞的情况,红色点代表标记了癌基因的ecDNA,绿色荧光Aurora是一种丝氨酸/苏氨酸蛋白激酶,参与细胞分裂有丝分裂。
3.实际统计不同ecDNA比例子细胞数量分布(柱状图)符合正态分布,和理论预测的结果(虚线)高度一致(fig.1c)
方法2:进一步验证上述结论,该研究设计了一个活细胞成像系统,能可视化细胞分裂时ecDNA的动态变化。(fig.1d,e)
1.用CRISPR-Case9技术在PC3前列腺癌细胞的ecDNA的MYC和PVT1之间插入TetO重复序列(fig.1d)
2.用PCR、Sanger测序、TetO-MYC双通道FISH确认了TetO重复序列的插入。(Extended Data Fig.3a-d)
3.随后表达融合绿色荧光蛋白(GFP)的TetR(TetR-GFP),可以和TetO结合,用于在细胞分裂过程中追踪ecDNA。(fig.1d)
4.用JF669 SNAP标签标记染色质上的组蛋白H2B,形成H2B-SNAP融合蛋白,追踪染色质。
5.通过动态影像,观察细胞分裂的过程中染色体和ecDNA进入子细胞的情况。
结果:可以看到染色质均匀分配到两个子细胞中,而ecDNA是随机分配的(fig.1e和Supplementary Video 1)
(2)通过计算机来模拟肿瘤细胞群中ecDNA的动态分布(Suplementary Information 2.1)
问题1:ecDNA的数量在肿瘤细胞群中是怎么动态分布的?
方法:研究通过计算机算法( Gillespie 算法 的 Metropolis Hastings 实现)来模拟肿瘤细胞群生长过程中单个细胞中ecDNA的数量变化
1.计算机运行这个算法,从1个带有1个或者k个ecDNA的肿瘤细胞开始(k代表该细胞系中每个细胞ecDNA的平均数),模拟该细胞逐渐生长分裂出一群细胞的过程中,ecDNA的数量在每个细胞中的动态变化。(fig.2a,c)
2.另外,该研究还对样本进行FISH镜下观察,统计每个细胞中的ecDNA数量并绘制曲线,与计算机模拟的结果进行比较。(fig.2a,c)
3.样本的选择:8组细胞系样本和2组临床样本进行算法的模拟(6个ecDNA+的肿瘤细胞系(其中2个细胞系有2种ecDNA,所以一共是8个组fig.2b),和2个患者肿瘤样本(GBM和NB fig.2d))。
结果:无论是模拟细胞系还是患者肿瘤样本,FISH的计数结果和计算机模拟的曲线都比较吻合。
(3)环境压力对ecDNA拷贝数的影响(fig.3)
问题1:药物对细胞的ecDNA拷贝数有没有影响?
背景:进化机制包括2种常见的理论:中性选择和正向选择。
1.中性选择:指某些基因变异或特征使生物体不对生物体的适应性产生显著影响,也就是这个突变对生物个体的生殖与生存既没有好处也没有坏处,不受自然选择的影响。
2.正向选择:指某些基因变异或特征使生物体在特定环境条件下具有更高的适应性,受自然选择的影响,这些变异或特点的频率会增加。
方法1:通过计算机算法和实验共同探究甲氨蝶呤对ecDNA拷贝数的影响,甲氨蝶呤是一种二氢叶酸还原酶抑制剂,是一种抗叶酸类抗肿瘤药。
1.通过CRISPR/Cas9技术在HAP1肿瘤细胞系(单倍体的人类髓性白血病细胞系)中构建含有DHFR(二氢叶酸还原酶基因)的ecDNA(Fig.3a)。实验开始时,约15%的细胞中有ecDNA,并且每个细胞只有1个拷贝的ecDNA。
2.实验验证:用甲氨蝶呤来处理细胞,使用微滴式数字PCR(ddPCR)来检测ecDNA拷贝数和染色体上的伤痕频率(scar:CRISPR切割染色体后留下的痕迹),观察细胞平均ecDNA拷贝数的变化情况。
3.算法预测:用算法预测这些细胞ecDNA的平均拷贝数变化情况。
结果:
1.ddPCR检测ecDNA平均拷贝数随时间的变化情况:观察到刚开始用CRISPR/Cas9诱导ecDNA产生后,无论有无HT(H:次黄嘌呤;T:胸腺嘧啶核苷)补充,随着细胞群规模的增大都会出现一段时间的ecDNA平均拷贝数下降,之后就会趋于稳定,细胞受到中性选择(fig.3b)。算法模拟fig.3b的实验,得到相同的结果(fig.3c)。
2.ddPCR检测Scar随时间的变化情况:无论有无HT补充,scar的频率都趋于稳定。(fig.3d)
3.ddPCR检测各浓度的甲氨蝶呤对细胞平均ecDNA拷贝数的影响:(fig.3e)
①在没有甲氨蝶呤处理的情况下,细胞平均ecDNA拷贝数会趋于稳定,细胞受到中性选择。
②在有甲氨蝶呤处理的情况下,细胞平均ecDNA拷贝数呈甲氨蝶呤剂量依赖性上升,表明含有DHFR的ecDNA的细胞在甲氨蝶呤的压力下有显著的选择优势,受到正向选择。算法模拟fig.3e的实验,得到相同的结果(fig.3f)
问题2:进一步探究含有癌基因的ecDNA是否会受到正向选择?
方法:通过CRISPR/Cas9利用gRNA靶向细胞基因组的不同位置,使该位点断裂,观察不同断裂点对细胞数量的影响(fig.3g)
1.选择结肠癌细胞系(COLO320-DM),设计靶向其ecDNA的基因间区、MYC基因上以及8号染色体的非扩增基因间区的gRNA。
2.通过慢病毒载体把Cas9和sgRNAs(single-guide RNAs)导入细胞系。
3.计算敲断ecDNA后细胞增殖的情况(细胞数量)和ecDNA的拷贝数的变化。
结果:
1.靶向8号染色体的细胞增殖情况和空白对照没有显著区别;靶向ecDNA的(基因间区和MYC区域)细胞的数量显著低于空白对照组。(fig.3h)
2.靶向ecDNA的细胞ecDNA拷贝数显著少于空白对照组(fig.3i)
(4)ecDNA在肿瘤快速适应环境压力时的作用
背景:(fig.4a)
1.肿瘤在遇到环境压力时(比如养料不足、有化疗药物的环境下),会通过调节细胞内基因组结构或其它未知的机制来抵御外界的压力,有些肿瘤调节能力很强,能快速适应环境,维持生存。
2.以前的研究显示,ecDNA的特性,比如没有着丝粒,能快速引起肿瘤细胞基因拷贝数的变化,可能是肿瘤快速适应环境压力的关键;而染色体上的基因是均匀进入子细胞的,因此无法改变基因拷贝数。
3.癌基因的扩增既可以出现在ecDNA上,也可以出现在染色体上,过去通过染色体核型分析,在肿瘤细胞的G显带标本上可以看到染色体上某段区域呈均匀无带纹的浅染区,这是基因重复扩增的产物,因此,我们又把染色体上基因的扩增称为HSR(均匀染色区)。
目的:通过实验,观察肿瘤细胞(ecDNA+/-)在不同环境压力下的生存能力,以及特定癌基因拷贝数的变化,证明ecDNA有助于肿瘤细胞的存活和耐药性的形成。
方法1:葡萄糖限制实验
1.样本选择:选择两个都有EGFRvIII扩增的同基因细胞系(即除了目标基因的区别,其它基因完全相同的2个细胞系)GBM39-EC细胞系(EGFRvIII在ecDNA上扩增)和GBM39-HSR细胞系(EGFRvIII在染色体上扩增,携带HSR(均匀染色区)的细胞)。
2.环境条件:和对照组相比(100% glucose),实验组培养环境中葡萄糖减少80%(20% glucose)。
3.观察指标:在低糖环境下,GBM39-EC和GBM39-HSR组的细胞数量和在正常环境下细胞数量的比例随着培养时间的变化;各细胞系的正常糖和低糖相比,细胞中癌基因平均拷贝数的变化。
结果:(fig.4b,c)
1.GBM39-EC在20%的葡萄糖限制条件下表现出很强的适应性,细胞数量并未显著减少(fig.4b),并且细胞扩增基因的平均拷贝数迅速减少(fig.4c),说明GBM39-EC在低糖环境下发生了基因组的适应性调整。
2.GBM39-HSR在20%的葡萄糖限制条件下表现出极强的敏感性,细胞生长显著受阻(fig.4b),且染色体上的扩增基因拷贝数没有发生变化(fig.4c),说明GBM39-HSR在低糖环境下无法快速调整基因组适应环境。
方法2:EGFR酪氨酸激酶抑制剂实验(厄洛替尼),靶向EGFR的抗肿瘤药
1.样本选择:同葡萄糖限制实验,GBM39-EC和GBM39-HSR。
2.环境条件:对照组:二甲基亚砜(DMSO);实验组:5μM厄洛替尼。
3.观察指标:实验组GBM39-EC和GBM39-HSR的细胞数量和对照组细胞数量的比例随着培养时间的变化;各细胞系的实验组和对照组相比,细胞中癌基因平均拷贝数的变化。
结果:(fig.4d,e)
1.GBM39-EC细胞:厄洛替尼治疗后,GBM39-EC细胞数量开始呈下降趋势,但仅在两周内数量就开始恢复,并产生了耐药性。另外,EGFRvIII基因的平均拷贝数也在不断变化,使得细胞快速适应药物,并且观察到这种拷贝数的变化是可逆的(先减少后增多)。
2.GBM39-HSR细胞:厄洛替尼治疗后,GBM39-EC细胞数量呈下降趋势,且观察期间数量没有回升,说明细胞对药物高度敏感,并未产生抗药性。EGFRvIII基因位于染色体上,平均拷贝数未发生变化。
结论:癌基因在ecDNA上才能迅速实现拷贝数变化,肿瘤细胞更能适应外界环境的压力。
方法3:患者肿瘤样本分析耐药性(EGFR酪氨酸激酶抑制剂拉帕替尼lapatinib)
1.样本选择:两位GBM患者的肿瘤样本。两位患者都经历了原发肿瘤的切除——肿瘤复发——拉帕替尼治疗——手术切除。
2.观察指标:比较两次手术切除样本中癌基因(EGFR)的细胞平均拷贝数(FISH)。
结果:在使用拉帕替尼治疗后,肿瘤中的EGFR拷贝数显著减少,表明ecDNA在实际的临床患者中也能够动态调整以适应治疗。(fig.4h)
方法4:其它肿瘤类型和其对应的靶向药(相同的实验设计,相同的观察指标)
1.MYCN扩增的神经母细胞瘤(NB)细胞系(TR14和CHP212)和患者肿瘤样本,化疗药物:长春新碱(fig.4f,g,i)。
2.CDK4扩增的NB细胞系(TR14),化疗药物:CDK4/6抑制剂:阿贝西利,哌柏西利(Extended Data Figs,7d,e和8)。
结论:观察到了和前面相同的结果,表明ecDNA在这些细胞中同样能够快速调整以适应化疗,产生耐药性。
总之,研究者通过实验展示了与染色体相比,ecDNA能在环境压力中为癌基因的扩增提供更大的异质性和灵活性,从而促进肿瘤细胞的存活和抗药性形成。
讨论:
该研究强调了ecDNA对癌症生物学的重要影响,提出了它在癌症发生和进化中的独特作用,并暗示未来的治疗策略可能需要针对ecDNA的特殊适应性来设计。
1. ecDNA在癌症中的独特作用:
拓扑结构的改变:ecDNA具有不同于染色体DNA的拓扑结构,这种独特的拓扑结构使其具有增强的染色质可及性,从而重塑基因调控网络,促进致癌基因的转录活动。换句话说,ecDNA更容易被转录因子和其他调控蛋白接触,激活或增强某些致癌基因的表达,从而推动癌症的发展。
ecDNA的高级空间结构:ecDNA不仅在单个层面上影响基因表达,多个ecDNA还能够聚集成“hub”,形成更复杂的高级组织结构。这种组织形式进一步增强了ecDNA在肿瘤发生中的致病作用。
2. ecDNA对达尔文进化原则的影响:
ecDNA以随机分离的方式遗传,这意味着它不遵循传统的孟德尔遗传规律。这种随机性带来了更多的变异性,增强了肿瘤细胞的进化潜力。ecDNA通过这种随机遗传,加速了肿瘤的进化进程,并且提高了肿瘤细胞适应环境压力(如药物治疗)的能力。传统疗法可能难以应对肿瘤细胞通过 ecDNA 快速调整基因表达和适应环境的能力。
3. 治疗挑战与未来方向:
由于ecDNA的随机性和快速适应性,单纯靶向扩增致癌基因的疗法可能难以在这种动态环境中取得持久效果。因此,治疗这些ecDNA阳性的癌症可能需要全新的策略,不仅针对致癌基因本身,还要针对ecDNA的独特适应性。例如,未来的治疗方案可能需要抑制ecDNA的形成、分离或调控机制,以降低肿瘤细胞的适应性和进化潜力。

汇报人:夏晓旭
导师:赵宇、任建君
审核:李朔、任建君



